Progetto sistemi intelligenti
Il questo progetto di sistemi intelligenti si vuole iniziare ad usare MatLab mettendo a confronto tre diversi algoritmi di algoritmi Unsupervised Learning:
- K-mean
- Fuzzy C Mean
- Self-organizing map (SOM)
Il risultato poi verrà messo a confronto attraverso "Davies-Bouldin Index" [link]
Primi passi con MatLab
I dati del progetto sono presi direttamente da Matlab, dal dataset cities. Il dataset include le valutazioni per 9 diversi indicatori della qualità della vita in 329 città degli Stati Uniti. Si tratta di:
- clima
- abitazioni
- salute
- criminalità
- trasporti
- istruzione
- arte
- svago
- economia
Una valutazione più è alta meglio è. Ad esempio, un punteggio più elevato per il crimine significa un tasso di criminalità più basso.
Il data set delle città contiene tre variabili:
- una matrice con le 9 categorie
- una matrice coi i 329 nomi di città
- la matrice di dati con 329 righe e 9 colonne
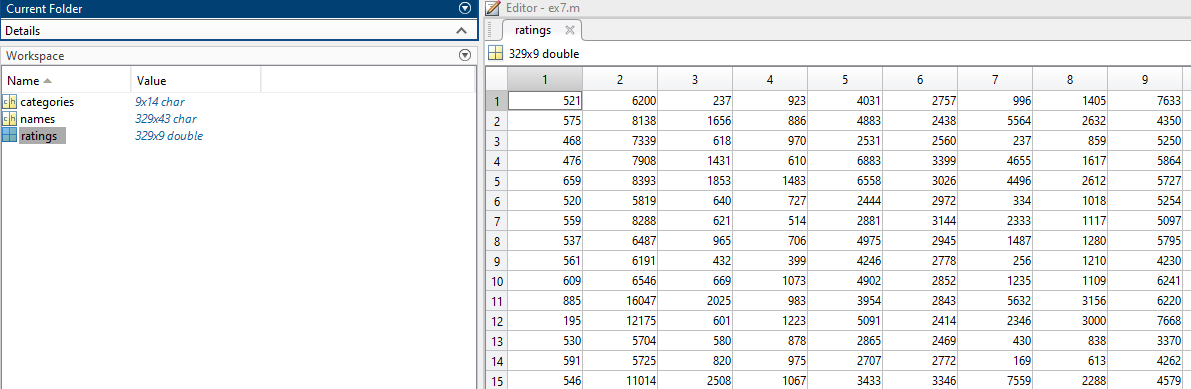
Una volta caricato il dataset è possibile mostrare i range che i valori assumono.
Questa immagine ci fa vedere che vi è una maggiore variabilità nelle categoria
"arte" e "abitazioni" rispetto alla categoria "criminalità" o "clima".
Se invece volessimo vedere tutte e 9 le categorie sotto forma di istogramma, per avere una maggiore comprensione di come i dati sono distribuiti
CodiceMatlab
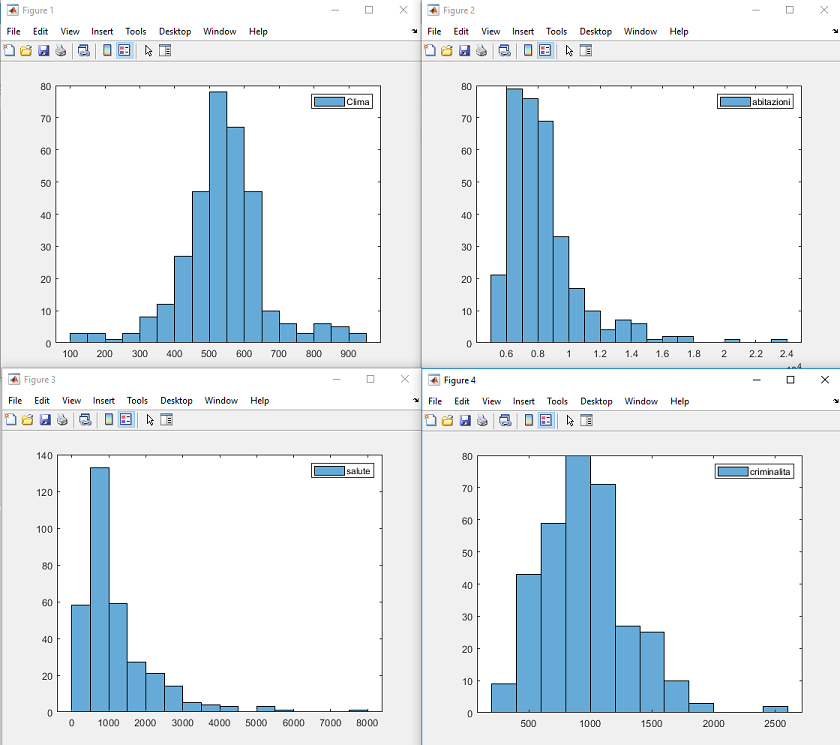
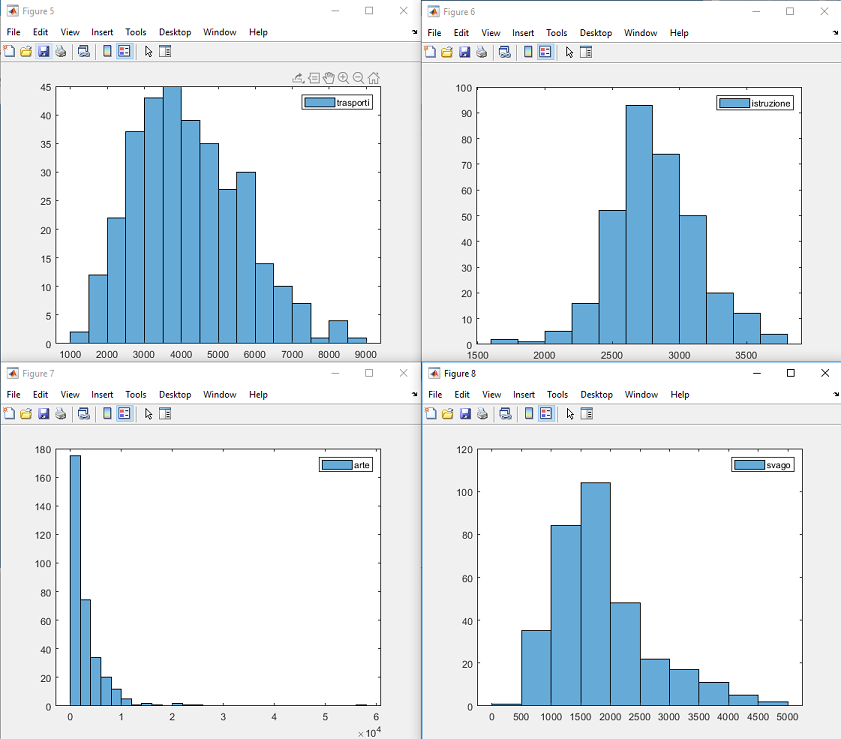
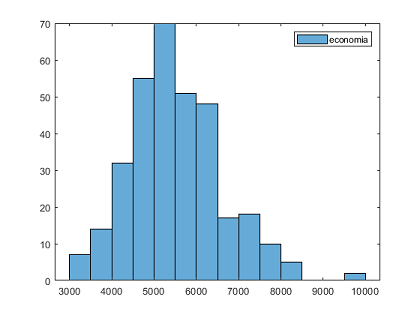
Possiamo subito vedere come le categorie "clima", "criminalita", "trasporti", "istruzione", ...
hanno una forma vagamente gaussiana: c'è un valore più popoloso, più ci si allontana meno più decresce la numerosità
Le categorie "abitazioni" e "arte" hanno un range di valori veramente ampio: fino a 10^4 valori
Per vedere la citta migliore e peggiore per ogni categoria, giusto per curiosità
city Worst Clima:Grand Forks, ND
city Best Clima:Oakland, CA
--------------------
city Worst abitazioni:Joplin, MO
city Best abitazioni:Stamford, CT
--------------------
city Worst salute:Glens Falls, NY
city Best salute:New York, NY
--------------------
city Worst criminalita:Wheeling, WV-OH
city Best criminalita:New York, NY
--------------------
city Worst trasporti:Houma-Thibodaux, LA
city Best trasporti:New York, NY
--------------------
city Worst istruzione:Pascagoula, MS
city Best istruzione:Philadelphia, PA-NJ
--------------------
city Worst arte:Sharon, PA
city Best arte:New York, NY
--------------------
city Worst svago:Gadsden, AL
city Best svago:Seattle, WA
--------------------
city Worst economia:Eugene-Springfield, OR
city Best economia:Midland, TX
Peggiore Clima:
 |
|
Migliore Clima:
 |
Estrazione/Selezione delle features
La bontà di una classificazione dipende in ugual misura
- dalla bontà delle features scelte (feature selection/ extraction)
- dalla bontà ed adeguatezza dell’algoritmo di classificazione rispetto al problema in esame
- Preprocessing
I criteri più semplici per definire la bontà delle features:
Ci dice qual è il “punto centrale” ma non caratterizza in modo esauriente una data popolazione
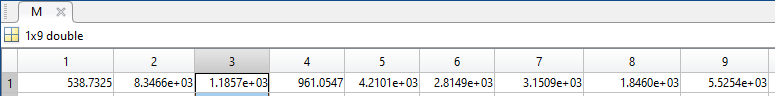
Dove A è la nostra matrice e M è il risultato. La media viene già fatta per colonna
M = mean(A)
Deviazione standard [link]
La deviazione standard è una misura dello spread e viene definita come la distanza media di un punto appartenente ad una data popolazione dal valore centrale.
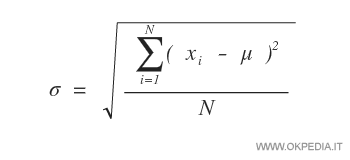
Covarianza [link]
In statistica e in teoria della probabilità, la covarianza di due variabili statistiche o variabili aleatorie è un numero che fornisce una misura di quanto le due varino assieme, ovvero della loro dipendenza.
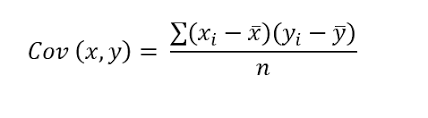 Wikipedia
Wikipedia

Per calcolare la covarianza, matlab ci offre gia la funzione.
Tuttavia per mostrare tutti i passaggi e impratichirsi con Matlab costriuamo passo passo la funzione Covarianza()
% ---------- CALCOLO MANUALE VARIANZA ------------
% citta da prendere, per l'esempio solo 5
valueToTake = 5;
% Prendo la prima colonna, fino al 5 valore
% Prendo la seconda colonna fino al 5 valore
% Infine combino i vettori in un unica matrice 5x2
A = [ratings(1:valueToTake,1) ratings(1:valueToTake,2)];
% Calcolo la media (per colonne)
M = mean(A);
% Creo un vettore di tutti zeri (con 5 righe)
X1 = zeros(size(ratings(1:valueToTake,1)));
% Combino questo vettore vuoto con la matrice per avere uno spazio per i
% calcoli
B = [A X1 X1 X1];
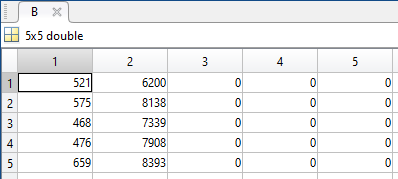
Ora computo valore ennesimo meno la media e salvo il valore nella rispettiva colonna
% Calcolo (valore 'ennesimo' - media) e lo salvo nella sua colonna
for i = 1:valueToTake
B(i,3) = B(i,1)-M(1,1);
end
t = M(1,2);
% Calcolo (valore 'ennesimo' della seconda features - media) e lo salvo nella sua colonna
for i = 1:valueToTake
B(i,4) = B(i,2)-M(1,2);
end
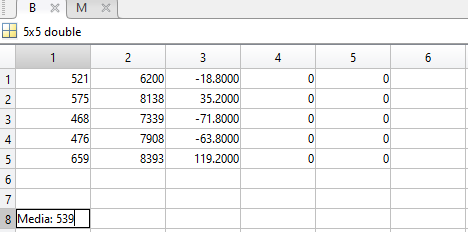
% Calcolo la moltiplicazione tra i 2 fattori e lo salvo nella sua colonna
for i = 1:valueToTake
B(i,5) = (B(i,3)) *(B(i,4));
end
% Calcolo covarianza
totColonna = 0;
for i = 1:valueToTake
t = (B(i,5));
totColonna = totColonna +t;
end
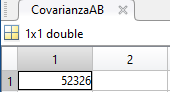
CovarianzaAB = totColonna / (valueToTake-1);
% Matlab
x1 = ratings(1:valueToTake,1);
x2 = ratings(1:valueToTake,2);
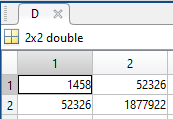
D = cov(x1,x2);
La matrice calcolata direttamente da MatLab
% ---------- FINE CALCOLO MANUALE VARIANZA ------------
La covarianza:
- con segno positivo: indica che le due dimensioni sono correlate in senso direttamente proporzionale (se una componente aumenta anche l’altra aumenta); la covarianza è una misura della dipendenza lineare tra le variabili
- pari a 0 indica che non c’è relazione nella variazione
- negativa variano in senso inversamente proporzionale (se una componente aumenta l’altra diminuisce)
In queste prime 5 citta il valore della covarianza è 52326 ovvero un valore positivo => se una componente aumenta l’altra aumenta.
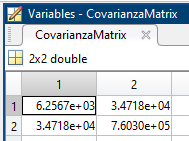
La matrice di covarianza per tutte le features è:
Wikipedia
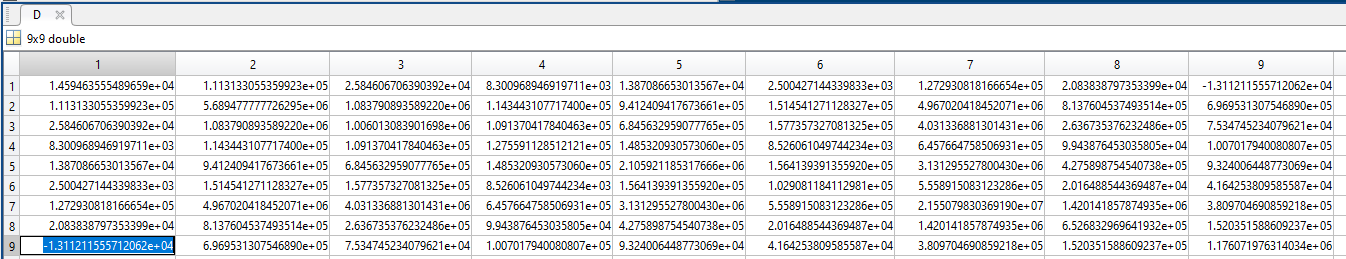
Come si può vedere tutte le features sono correlate tra loro con valori positivi, come giustamente si poteva immaginare.
L'unico valore negativo è nella correlazione climate e economics che suggerisce che il bel tempo sfavorisce l'economia (?)
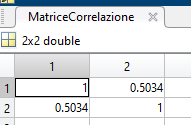
Matrice di correlazione [link]
Dalla matrice di co-varianza si può calcolare la matrice di correlazione R che direttamente riporta il grado di correlazione delle features.
Gli elementi di R sono legati agli elementi della matrice di covarianza dalla relazione:
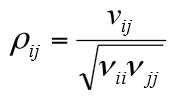
Vij Elementi della matrice di covarianza
Vii e Vjj Elementi della diagonale della matrice di covarianza di posizione ii e jj ovvero varianze delle feature i e j rispettivamente
Per calcolare la matrice di correlazione, matlab ci offre gia la funzione.
Tuttavia per mostrare tutti i passaggi e impratichirsi con Matlab costriuamo passo passo la funzione matriceCorrelazione()
Coordinate dei punti delle 5 città prese come esempio
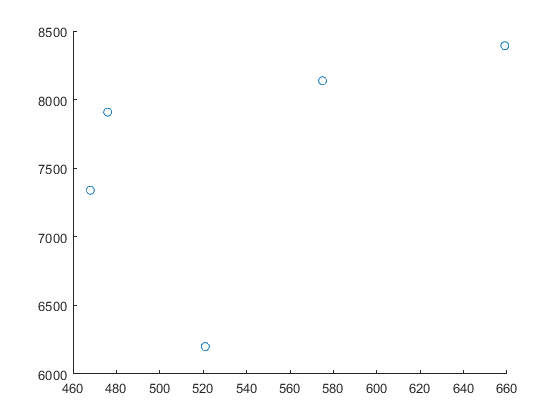
% ---------- CALCOLO MANUALE MATRICE CORRELAZIONE ------------
% citta da prendere, per l'esempio solo 5
valueToTake = 5;
% Prendo la prima colonna, fino al 5 valore
% Prendo la seconda colonna fino al 5 valore
x1 = ratings(1:valueToTake,1);
x2 = ratings(1:valueToTake,2);
A = [x1 x2];
% stampo il grafico
% scatter(x1,x2);
% Calcolo la media delle 2 features
Mvector = mean(A);
M = [Mvector(1,1); Mvector(1,2)];
% Creo l'oggetto cell dove mi posso salvare i valori intermedi dei calcoli
CellArrey = cell(valueToTake, 3);
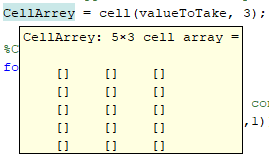
%Calcolo vero a proprio
for i = 1:valueToTake
% 1) creo la matrice (2x1) con i 2 valori
temp_mat = [x1(i,1); x2(i,1)];
CellArrey{i,1} = temp_mat;
% 2) salvo la differenza tra il valore del punto attuale e la media
CellArrey{i,2} = minus(temp_mat,M);
G = minus(temp_mat,M);
%3) Mi calcolo la matrice appena trovata per se stessa trasposta
Gtrasposta = G.';
Step3 = G * Gtrasposta;
CellArrey{i,3} = Step3;
end
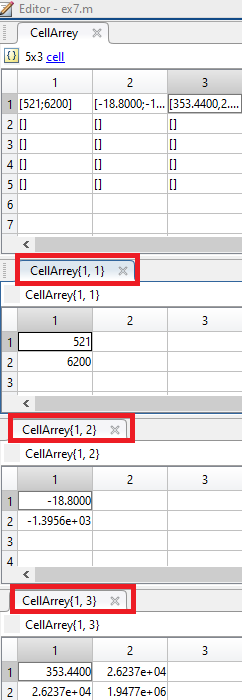
Nella cella 1.1 ci sono i valori del punto
Nella cella 1.2 ci sono i valori del punto meno la media delle features
Nella cella 1.3 c'è il risultato della cella 1.2 per la sua trasposta
% Creo la matrice per il calcolo finale
CovarianzaMatrix = [0,0;0,0];
% Calcolo la matrice di covarianza, sommando tutti i valori parziali e
% dividendo il risultato per n-1
% Calcolo la matrice di covarianza, sommando tutti i valori parziali
for i = 1:valueToTake
% Mi salvo temporaneamente la matrice salvata nell'oggetto CellArrey
tempCell = CellArrey{i,3};
% Mi salvo la somma parziale progressiva di tutti i valori:
% Riga=1 e Colonna=1
CovarianzaMatrix(1,1) = CovarianzaMatrix(1,1) + tempCell(1,1);
% Riga=1 e Colonna=2
CovarianzaMatrix(1,2) = CovarianzaMatrix(1,2) + tempCell(1,2);
% Riga=1 e Colonna=2
CovarianzaMatrix(2,1) = CovarianzaMatrix(2,1) + tempCell(2,1);
% Riga=2 e Colonna=2
CovarianzaMatrix(2,2) = CovarianzaMatrix(2,2) + tempCell(2,2);
end
% dividendo il risultato per n-1
CovarianzaMatrix(1,1) = CovarianzaMatrix(1,1) / (valueToTake-1);
CovarianzaMatrix(1,2) = CovarianzaMatrix(1,2) / (valueToTake-1);
CovarianzaMatrix(2,1) = CovarianzaMatrix(2,1) / (valueToTake-1);
CovarianzaMatrix(2,2) = CovarianzaMatrix(2,2) / (valueToTake-1);
MatriceCorrelazione = [0,0;0,0];
% Calcolo la matrice di correlazione
% Si prendono gli indici i e j e si calcola per ogni cella il valore
% con la formula (ij)/((ii)*(jj))^1/2
for i = 1:2
for j = 1:2
MatriceCorrelazione(i,j) = CovarianzaMatrix(i,j) / sqrt(CovarianzaMatrix(i,i) *CovarianzaMatrix(j,j));
end
end
%% ---------- FINE CALCOLO MANUALE MATRICE CORRELAZIONE ------------
% Matlab
E = corr(ratings,ratings);
Prendendo tutti i 329 punti sia con matlab che con la funzione manuale troviamo lo stesso risultato
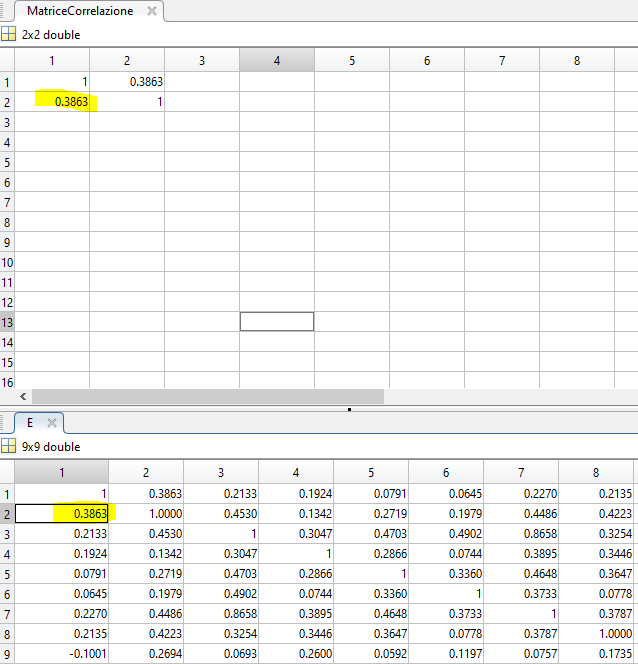
Kmeans
Kmeans su Wikipedia
Descrizione generale
L'algoritmo K-means è un algoritmo di clustering partizionale che permette di suddividere un insieme di oggetti in K gruppi sulla base dei loro attributi. Si assume che gli attributi degli oggetti possano essere rappresentati come vettori, e che quindi formino uno spazio vettoriale.
L'obiettivo che l'algoritmo si prepone è di minimizzare la varianza totale intra-cluster. Ogni cluster viene identificato mediante un centroide o punto medio. L'algoritmo segue una procedura iterativa. Inizialmente crea K partizioni e assegna ad ogni partizione i punti d'ingresso o casualmente o usando alcune informazioni euristiche.
Quindi calcola il centroide di ogni gruppo. Costruisce quindi una nuova partizione associando ogni punto d'ingresso al cluster il cui centroide è più vicino ad esso. Quindi vengono ricalcolati i centroidi per i nuovi cluster e così via, finché l'algoritmo non converge.
Kmeans con matlab
Il primo esperimento che è possibile fare con il KMean è prendere solamente le prime 2 features (clima e abitazione) e dividere il dati in 2 cluster.

Il risutato è:
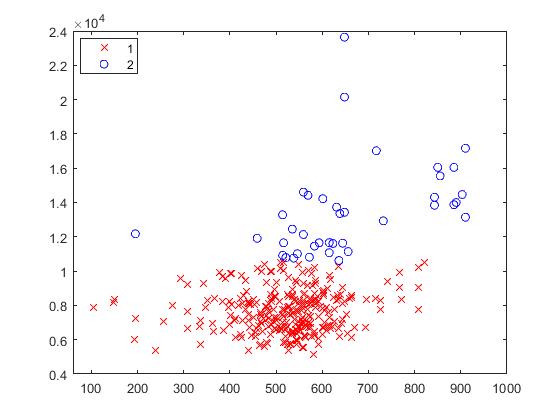
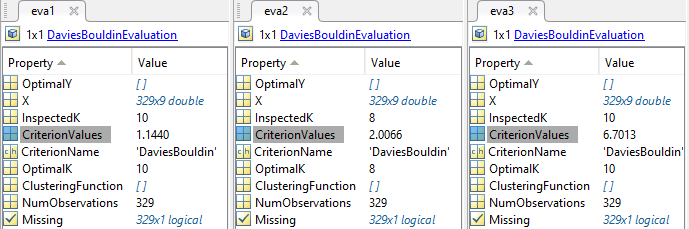
Matlab mette a disposizione la funzione evalclusters() che calcola, grazie al "Davies-Bouldin index", di stimare in quanti cluster sarebbe meglio dividere i dati.
Davies-Bouldin index
- rapporto tra la somma delle distanze medie tra gli elementi e il centro di due cluster e la distanza (dal centro) tra i due cluster
- sommato per ogni coppia di cluster
- va minimizzato (significa cluster ben compatti con centri distanti tra loro)
Calcolando il miglior numero di cluster:
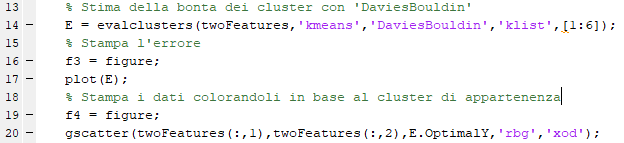
L'errore in base al n dei cluster è:
Il risultato dividendo la popolazione in 5 cluster:
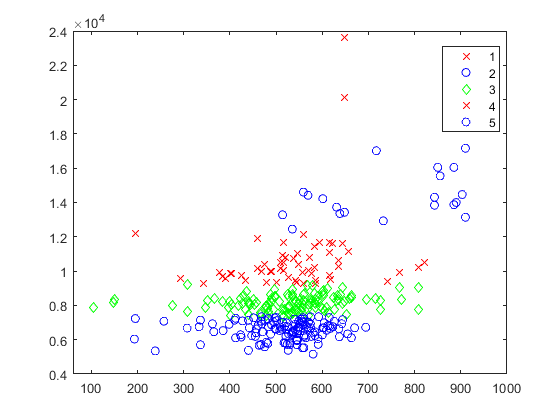
Per continuare ad esercitasi con Matlab e il Kmeans, il secondo esperimento svolto è stato il confronto tra:
- Il KMeans analizzando tutte e 9 le features
- Il KMeans su ogni singola features, poi la media dei valori ottenuti
- Il KMeans su ogni singola features, poi contando "la frequenza delle label"
Variando k e misurando il grado di similarità tra i 3 modi di dividere i dati sempre con "Davies-Bouldin index"
é possibile prendere la label più frequente tra le features, perchè i dati dopo essere stati clusterizzati sono stati ordinati per valore
il primo cluster ha sempre i valori più bassi, così a crescere fino al kappesino cluster che raggruppa i valori più grandi
- Inizialmente calcolo il KMeans per ogni featurer dividendo i dati in 10 classi:
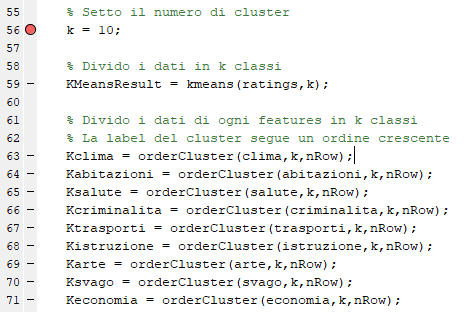
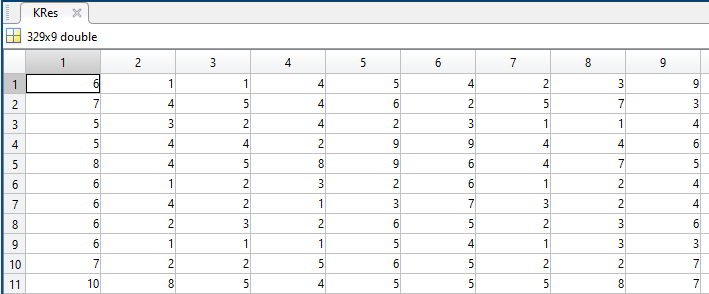
- Compongo una matrice con tutte le features:

- Ogni colonna rappresenta una features, mentre nelle righe si vede in quale cluster è stato classificato il valore (da 1 a 10)
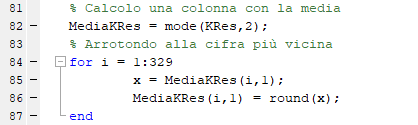
- Calcolo il cluster che viene utilizzato di più, e setto come etichetta questo valore

- Calcolo la media tra i cluster, arrotondando alla cifra intera più vicina
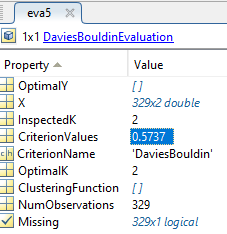
Infine viene calcolato, sempre con "Davies-Bouldin index", quanto i dati sono stati clasterizzati . Più l'indice è vicino allo zero meglio è
Le variabili nell'esperimento sono quindi: K, Kmeans originale, KMeans tra le features e prendendo la media e l'etichetta con il valore maggiore

Il risultato è per k = 10:
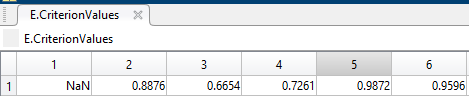
Variando k (e prendendo 2 volte i valori) i riusultati sono:
| K |
Kmeans |
Kmeans media |
Kmeans frequenza > |
| 2 |
0.88 |
1.17 |
1.17 |
| 2 |
0.57 |
1.23 |
1.23 |
| 3 |
0.66 |
1.25 |
1.61 |
| 3 |
0.66 |
1.34 |
1.71 |
| 4 |
0.75 |
1.78 |
3.49 |
| 4 |
1.025 |
1.36 |
2.7 |
Se per una persona leggere i valori separati di ogni features può essere una soluzione più comprensibile, questo modo di raggruppare i dati non raggiunge gli stessi livelli ottenibili con i Kmeans.
Facendo analizzare tutto il dataset con la funzione nativa di matlab evalclusters(), il risultato è sempre 3 cluster
! Matlab ogni volta classifica i cluster in modo random, non si possono confrontare le etichette tra le varie features se non vengono ordinate
è stata quindi implementata una funzione che ordina le etichette dei cluster in base al loro valore, quindi identificando nei cluster con etichetta '1' i valori più piccoli,
con etichetta '10' quelli con valore maggiore
Si può fare questo ordine perchè nel dataset tutte le features seguono lo stesso schema (dal peggiore al migliore)
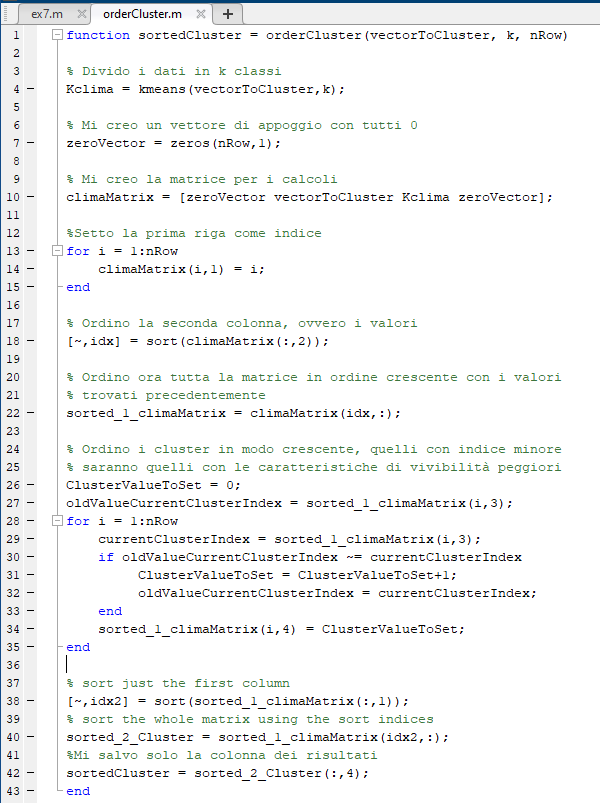
La funzione passo passo è:
1) Divido i dati in k classi
Kclima = kmeans(vectorToCluster,k);
2) Creo una matrice con un indice crescente nella prima colonna, il valore nella seconda colonna e nella terza colonna l'etichetta che è stata messa al cluster
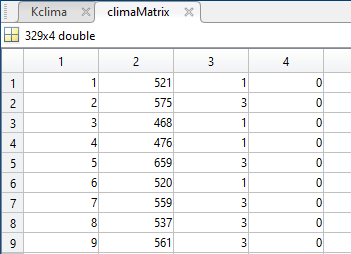
3) Ordino per il valore della features
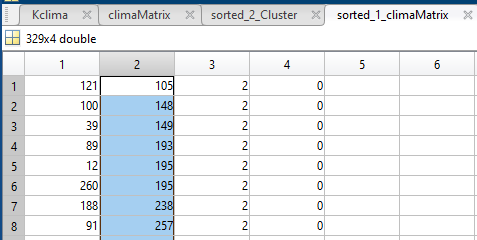
4) Rinomino le etichette, prima i valori più "piccoli" erano etichettati con '2', ora con '1'
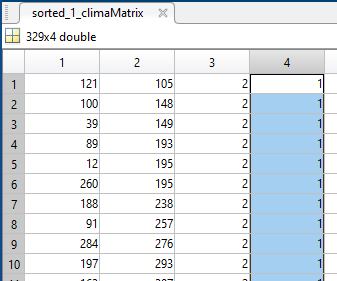
5) Riordino di nuovo in base all'indice creato all'inizio, rimettendo nell'ordine di partenza le varie città
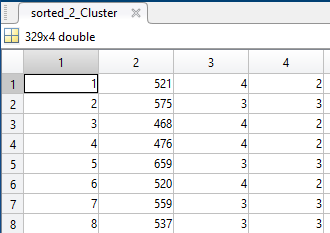
6) Prendo solamente la 4 colonna con le etichette "ordinate" del cluster
Fuzzy c-means clustering
Fuzzy c-means su Wikipedia
Fuzzy c-means su matlab
Descrizione generale
Dato un insieme di dati, detto dataset, lo scopo del clustering è quello di identificare raggruppamenti naturali di questi punti, per cercare di ottenere una concisa rappresentazione del comportamento del sistema.
Fuzzy C-Means (FCM) è la tecnica di clustering che utilizzata per il secondo esperimento: tale approccio metodologico, prendendo ispirazione direttamente dalla Fuzzy Logic, è una tecnica di data-clustering in cui, per ciascun punto del set di training si determina un certo grado di membership per ciascuno dei cluster che vengono individuati.
Sostanzialmente, dati D punti, l’obiettivo è suddividerli in N partizioni distinte; l’aspetto particolare e innovativo rispetto al clustering standard è che questo approccio consente di determinare quali sono i gradi di appartenenza (livelli di sfumatura) dell’i-esimo punto rispetto a ciascuna delle partizioni considerate.
L’algoritmo opera assegnando in maniera casuale un certo grado di membership a ciascun punto i rispetto ad ognuno dei j cluster e ricavando di conseguenza una certa distribuzione spaziale iniziale dei centri di massa delle N partizioni che si vogliono ricavare.
Attraverso un procedimento iterativo, la funzione muove dinamicamente i baricentri verso la localizzazione ottima, ovvero andando a minimizzare un funzionale di costo che rappresenta la somma delle distanze euclidee di ciascun punto da ciascun centro di massa, opportunamente pesate col corretto grado di membership.
Definizione di "Fuzzy c-means" da "L’algoritmo Fuzzy C-Means clustering come tecnica automatica per l’individuazione di anomalie di mercato: un caso studio"
Fuzzy c-means con matlab
Il primo esperimento che è possibile fare con il Fuzzy c-means è prendere solamente le prime 2 features (clima e abitazione)
e dividere il dati in 2 cluster, così da poterlo confrontare con i KMeans
Purtroppo Matlab mette non mette a disposizione, usando come algoritmo il "Fuzzy c-means", la funzione evalclusters(),
bisogna quindi variare manualmente il K per trovare il K che minimizza l'errore
Il codice ovviamente non è uguale a quello per il KMean, infatti la funzione che calcola il "Fuzzy c-means"
ci restituisce un grado di appartenenza alla feature, quindi per poter valutare tramite il "Davies-Bouldin index"
bisogna etichettare il dato con la label del centroide che più si avvicina.
Ovvero viene trasformando l'output da dato "continuo" (0.341, 0.612, ... ) in dato "discreto" (1,2,3...)
Il codice è:

Il risultato della funzione "Fuzzy c-means" è una matrice con le coordinate dei 2 centroidi
e una matrice che mi indica, per ogni città, la sua vicinanza al centroide 1 oppure 2
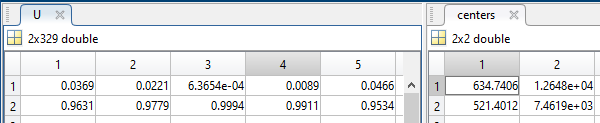
Per calcolare i 2 centroidi Matlab ci fa vedere anche tutte le varie iterazioni:
Iteration count = 1, obj. fcn = 1129490813.148721
Iteration count = 2, obj. fcn = 902502247.859216
Iteration count = 3, obj. fcn = 787330144.347303
Iteration count = 4, obj. fcn = 660063738.830454
...
Iteration count = 55, obj. fcn = 567271815.830051
Iteration count = 56, obj. fcn = 567271815.830043
Con sole 2 features è possibile facilmente stampare il grafico
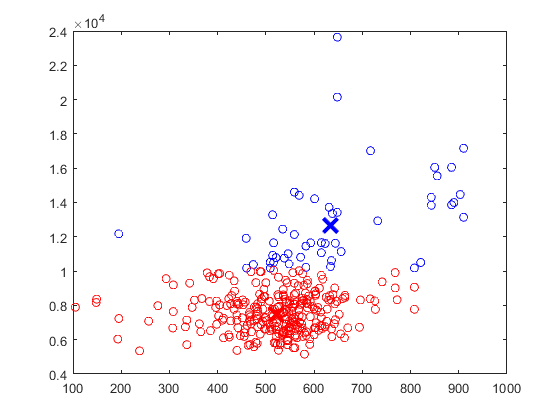
Infine viene calcolato, sempre con "Davies-Bouldin index", quanto i dati sono stati clasterizzati, ovviamente una volta che si è attribuita un etichetta (a seconda della max verosimiglianza) ad ogni dato
Per invece confrontare i 2 algoritmi con k che varia da 2 a 10 su tutti i dati:
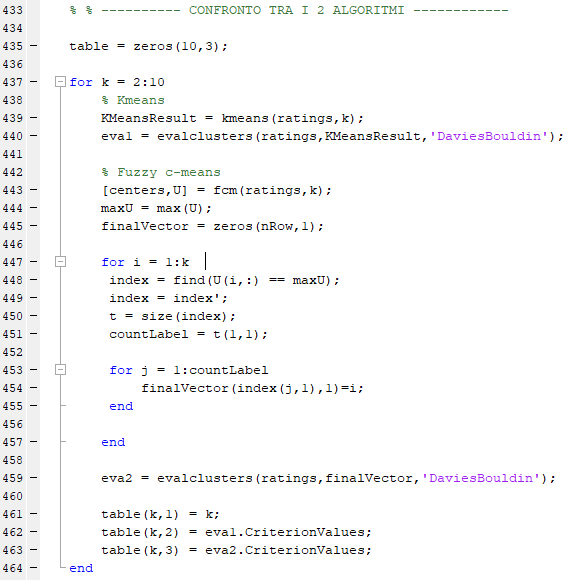
Il risultato è:
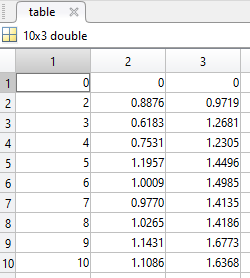
Nella prima colonna c'è il valore di K; nella seconda e nella terza il valore dell'indice "Davies-Bouldin" analizzando il dataset con Kmeans e Fuzzy c-means
Si può notare che il KMeans riesce meglio a raggruppare i dati.
Ovviamente la gradazione che il Fuzzy c-means ci restituisce non viene utilizzata nella sua complessita.
Quindi anche se è possibile fare un confronto bisogna sempre tenere conto di questo aspetto non secondario
Self-Organizing Map
Self-Organizing Map su Wikipedia
Descrizione generale
Le self-organizing map (SOM) sono un tipo di organizzazione di processi di informazione in rete analoghi alle reti neurali artificiali.
Sono addestrate usando l'apprendimento non supervisionato per produrre una rappresentazione dei campioni di training in uno spazio a bassa dimensione preservando le proprietà topologiche dello spazio degli ingressi. Questa proprietà rende le SOM particolarmente utili per la visualizzazione di dati di dimensione elevata
Le self-organizing map sono reti neurali a connessioni laterali dove i neuroni di uscita sono organizzati in griglie di bassa dimensione (generalmente 2D o 3D).
Ogni ingresso è connesso a tutti i neuroni di uscita. A ogni neurone è associato un vettore dei pesi della stessa dimensione dei vettori d'ingresso. La dimensione del vettore d'ingresso è generalmente molto più alta della dimensione della griglia di uscita. Le SOM sono principalmente usate per la riduzione della dimensione piuttosto che per l'espansione.
Self-Organizing slide Elearning
Le reti di Kohonen seguono l’organizzazione dei modelli competitive ma aggiungono una caratteristica specifica che fa sì che le relazioni topologiche di vicinanza dello spazio degli ingressi vengono riprodotte nello strato di uscita.
La rete Self Organising Feature Map (SOFM) o mappa di Kohonen ha una topologia a due livelli. Il livello superiore è costituito da uno strato bidimensionale (mappa) di neuroni la cui posizione nella mappa è identificata dalle sue coordinate.
Cambia, quindi la regola di apprendimento che diventa
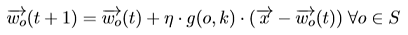
dove g(o,k) è una funzione decrescente della distanza tra il neurone k vincitore ed il neurone o considerato, mentre S è l’insieme dei neuroni. L’aggiornamento dei pesi viene così modulato in funzione della distanza della loro posizione da quella del neurone vincitore.
La funzione g dovrà avere valori maggiori per posizioni o vicine a k.
Una scelta usuale per g è quella della funzione gaussiana. Un’altra espressione per la funzione g è il Laplaciano della gaussiana. Tale funzione assume la forma di “cappello messicano”.
Da questo learning collettivo, per il quale i neuroni appartenenti ad un dato vicinato vengono aggiornati in modo decrescente rispetto al neurone vincente, si ha che neuroni vicini riconoscono input simili, ovvero input simili verranno mappati in neuroni vicini.
Le relazioni di distanza nello spazio n-dimensionale degli ingressi sono rappresentate approssimativamente da relazioni di distanza sulla mappa neurale bidimensionale. I concetti di “distanza” e conseguentemente di intorno si definiscono all’interno del reticolo discreto come approssimazione della distanza euclidea nel continuo.
Al termine del processo di learning, lo spazio n-dimensionale di input verrà mappato in modo “approssimato” dallo spazio bidimensionale della mappa di neuroni.
La topologia inerente lo spazio degli ingressi viene quindi preservata dalla rete detta appunto “topology preserving”.
Anche in questa caso è possibile introdurre nel modello due variazioni possibili alla regola di aggiornamento dei pesi:
- la prima consiste nel rendere il raggio dell’intorno interessato all’aggiornamento decrescente nel tempo
- la seconda variazione consiste nel diminuire il valore del learning rate all’avanzare dell’apprendimento.
Riassumendo, il processo di apprendimento self organizing consiste di 4 fasi:
- Inizializzazione random dei pesi;
- Competizione, ossia calcolo del neurone vincitore;
- Cooperazione, ossia il neurone vincitore determina la localizzazione di un intorno topologico nello spazio dei neuroni attivati;
- Fase adattiva che regola l’aggiornamento dei pesi con riduzione dell’intorno topologico e con diminuzione del tasso di apprendimento.
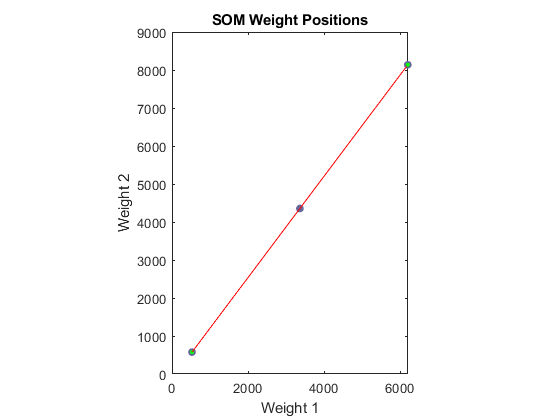
Self-Organizing Map con matlab
Il codice è per testare le prime 2 features con la Self-Organizing è
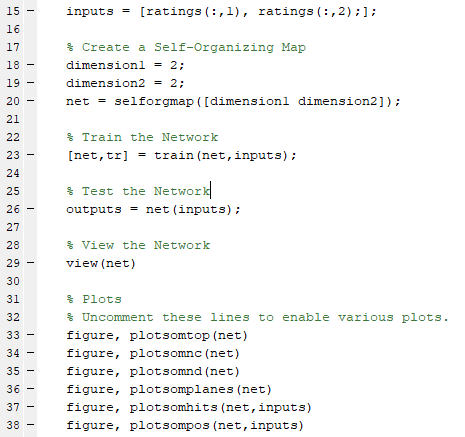
Il sistema sarà quindi organizzato:
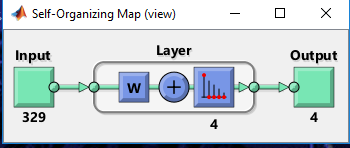
Matlab però permette di usare anche un interfaccia grafica

Selezionando semplicemente le prime 2 features (clima e città) matlab ci fornisce in automatico questi grafici come output:
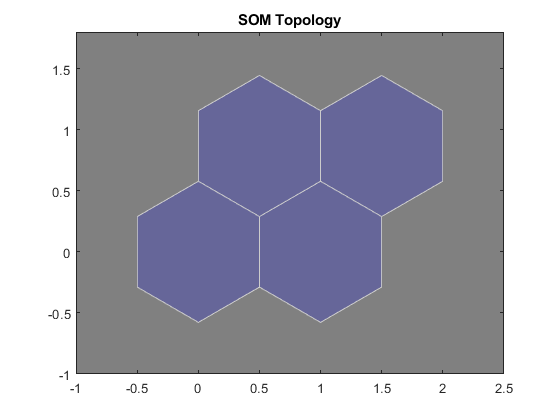
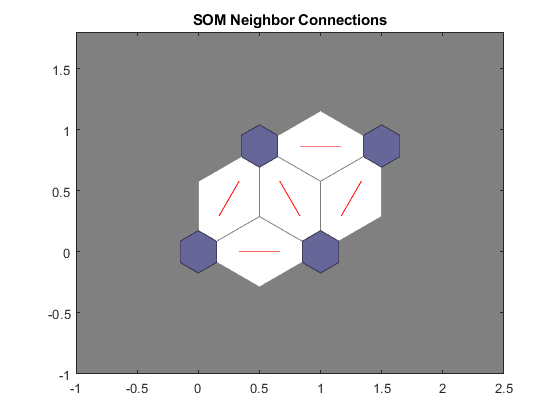
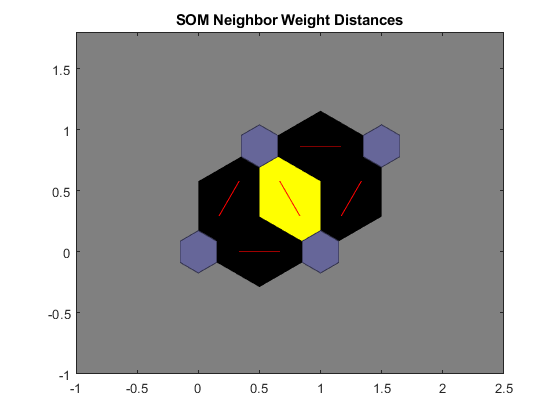
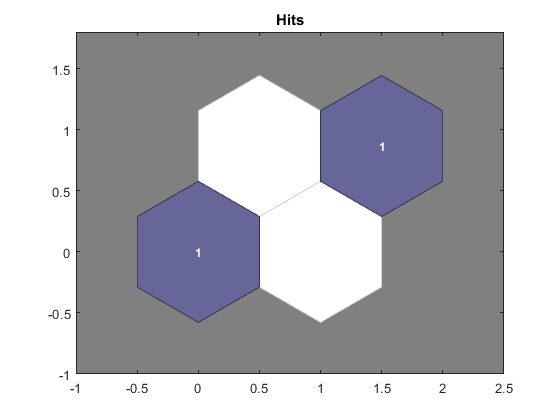
Esperimenti di laboratorio
JuzzyOnline - Gradimento del tuo lavoro
Growing Neural Gas
Varie & codice Matlab
Visualizza il codice on line del progetto
Implementazione da zero KMeans
Riepilogo comandi Matlab utili:
- scatter stampa i "pallini" - no quindi il comando plot()
- cell(n, m); salvare "oggetti" direttamente nella matrice link
- M = mode(A,2) find the most frequent value of each row link
- sort ordina in base al valore di una colonna link
// Input matrice 329x9
// Idea -> prendo la prima colonna e la faccio visualizzare come istogramma
To refer to multiple elements of an array, use the colon operator, which allows you to specify a range of the form start:end. For example, list the elements in the first three rows and the second column of A:
f1 = figure; // Creo la nuova figura
clima = ratings(1:329,1); // Creo un vettore dalla matrice
figClima = histogram(clima); // Creo l'istogramma della vettore
legend('Clima'); // Mostro la legenda
Per stampare un istogramma (con la legenda)
figClima = histogram(clima);
legend('Clima');
[M,I] = min(A) // Mi salva in M il è il valore minimo e I è l'indice del valore minimo
[climaMin,indexMinClima] = min(clima);
[climaMax,indexMaxClima] = max(clima);
cityWorstClima = names(indexMinClima,:);
cityBestClima = names(indexMaxClima,:);
disp(['city Worst Clima:', cityWorstClima]);
disp(['city Best Clima:', cityBestClima]);
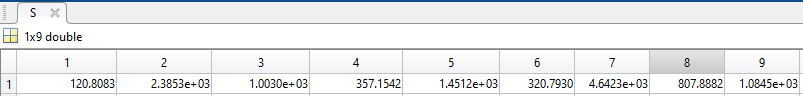
S = std(A) returns the standard deviation of the elements of A along the first array dimension whose size does not equal 1.
S = std(A)
Link:
- StatQuest: Principal Component Analysis (PCA), Step-by-Step
https://www.youtube.com/watch?v=FgakZw6K1QQ
https://www.youtube.com/watch?v=g-Hb26agBFg
- Dispese
http://www.oacn.inaf.it/~brescia/documents/ASTROINFOEDU/brescia-L6-MLunsupervised_partII.pdf
- Clustering Evaluation
https://www.cs.upc.edu/~bejar/URL/material/04-Validation.pdf